

聚合酶链式反应(PolymeraseChainReaction,PCR),是用DNA聚合酶在体外对特定的DNA片段进行快速扩增的方法。
该方法特异性高、敏感性强、操作简单、快速,不通过活细胞,在数小时内可使几个拷贝的模板序列甚至一个DNA分子扩增10~109倍,现已广泛应用到分子生物学研究的各个领域。
知名期刊PCR实验投稿要求
1. Nature系列期刊
原文要求:
"qPCR data must include primer sequences (with validation), amplification efficiency (90–110%), Ct values for all replicates, and MIQE guideline compliance. Normalization must use validated reference genes."
来源:Nature Methods Guidelines
关键要求:
引物序列+验证数据(如BLAST结果)
扩增效率(标准曲线法计算)
遵守MIQE指南
内参基因稳定性报告(如geNorm分析)
2. Cell系列期刊
原文要求:
"Provide raw Ct values, melt curves for specificity, primer sequences, and statistical methods. Biological replicates must be clearly stated."
来源:Cell Author Instructions
关键要求:
原始Ct值表格
熔解曲线证明单一峰值
生物重复≥3次(非技术重复)
3. Journal of Biological Chemistry (JBC)
原文要求:
"qPCR studies require amplification plots, primer efficiencies (R²≥0.98), and gel electrophoresis of PCR products. Data from ≥3 independent experiments must be shown."
来源:JBC Editorial Policies
关键要求:
扩增曲线图+产物电泳图
线性回归系数R²≥0.98
4. 《细胞与分子免疫学杂志》
原文要求:
"RT-qPCR需提供内参基因名称、引物序列、退火温度及循环数。数据须基于≥3次独立实验,并标注统计学差异(*p<0.05, **p<0.01)。"
参考链接:投稿指南
关键要求:
独立实验次数明确
统计学显著性标注
5. 《中国细胞生物学学报》
原文要求:
"PCR数据需包含引物序列、退火温度、产物大小及电泳图。定量结果需提供原始Ct值及标准化方法。"
来源:作者指南
关键要求:
凝胶电泳验证产物特异性
原始数据可追溯(如Excel表格)
6. 期刊特殊要求对比表

1. 实验设计阶段
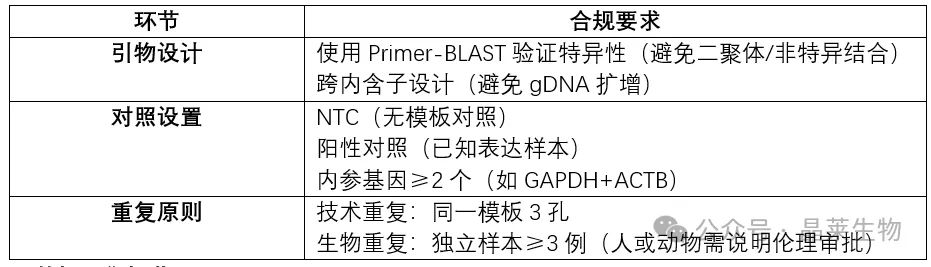
2. 数据采集规范
必需参数:
① 扩增效率:90–110%(计算方式:*E = [10^{(−1/slope)} − 1]×100%*)
② R²值:≥0.98(标准曲线线性度)
③ Ct值变异系数(CV):技术重复间<0.5
3. 核心经验总结
数据可重现性:
① 保存所有原始数据(仪器导出文件、凝胶图原始照片)。
② 记录试剂批号与仪器型号(如Applied Biosystems 7500)。
MIQE清单自查:
① 使用MIQE指南清单核对26项必填信息。
② 重点检查:RNA质量(RIN≥7)、cDNA合成方法(含gDNA去除)。
中文期刊特殊要求:
① 提供伦理委员会批准编号(涉及人类/动物样本)。
② 引物序列需标注合成公司。
1. 常见问题及解决方法
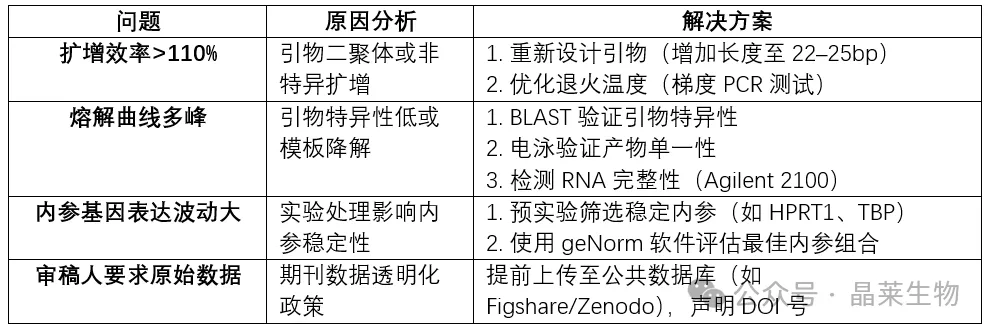
2. 投稿前终极检查清单
① 引物信息:序列、浓度、退火温度、扩增产物大小
② 验证文件:熔解曲线图、标准曲线图、电泳图
③ 统计数据:ΔΔCt计算过程、ANOVA分析结果、个体数据点
④ 伦理声明(如适用):批准编号、遵守《赫尔辛基宣言》
重要提示:Nature/Cell期刊可能要求提供qPCR仪原始输出文件(如.eds或.rdml格式),建议使用RDML标准格式保存数据。
通过严格执行上述指南,可显著提升稿件接收率,并确保实验结果的可重复性与学术诚信。
精准高效 · 一站式PCR实验解决方案
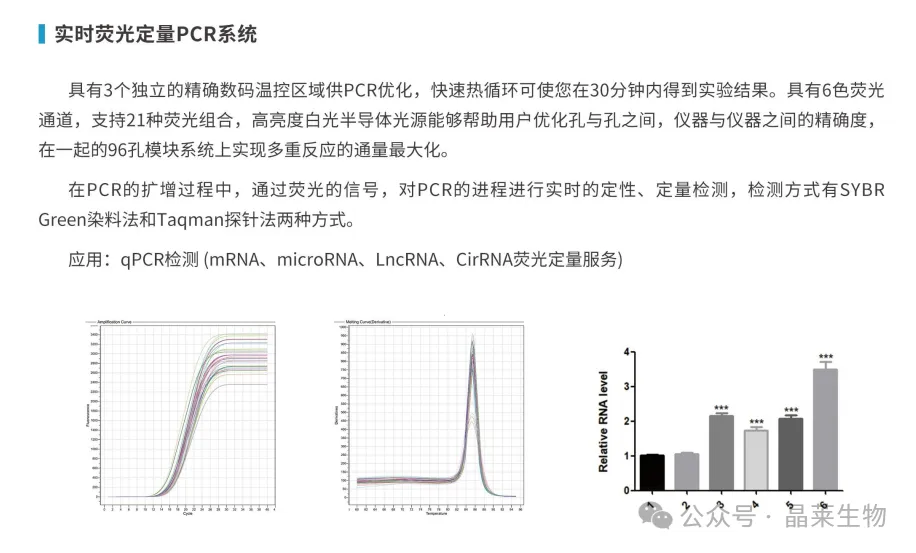
晶莱生物提供全流程合规PCR服务,确保数据全球期刊认可(Nature/Cell/JBC等)。
1. 引物设计(Primer-BLAST验证+跨外显子优化)
2. 三重质控:扩增效率≥95%|R²>0.99|内参基因稳定性分析(geNorm)
3. 技术全覆盖:从基础qPCR到前沿单分子检测
>> 立即联系我司技术顾问,解锁您的精准数据生产力!
关于晶莱
